Paso 3: Simulación en CanDo
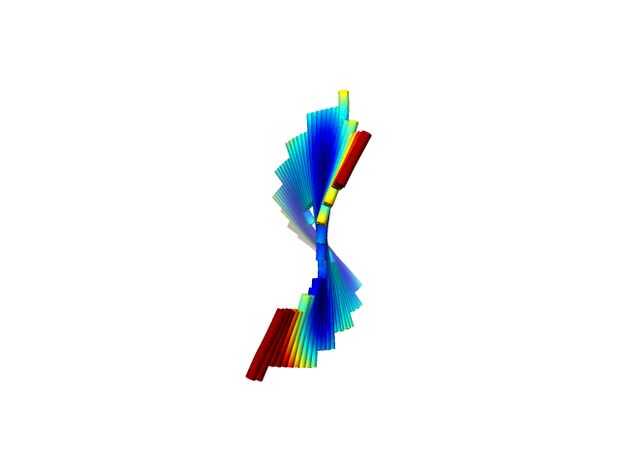

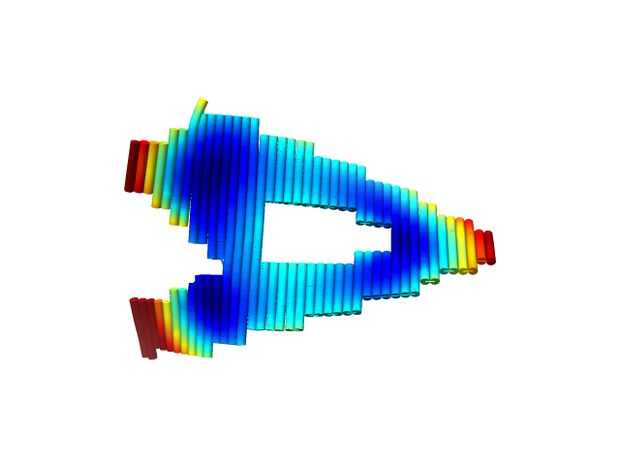
Una vez que habíamos diseñado y visualizado la nanoestructura, queríamos simular el origami para verificar que nuestro diseño era robusto. Origami de ADN es un material muy flexible, los crossovers en el diseño se utilizan para hacer la estructura más rígida. Hay un montón de diferentes maneras a los crossovers y para decir si una estructura tiene piezas rígidas que utiliza la aplicación web de CanDo para simular nuestro diseño.
La solución 3D forma y flexibilidad de nanoestructuras de ADN son predichos basado en el supuesto de que la respuesta mecánica de la doble hélice de ADN se aproxima bien por una barra elástica homogénea con módulos axiales, torsión y flexión que se han medido experimentalmente usando una variedad de directo, una sola molécula, así como técnicas de conjunto [8]. Cruces de doble cadena se modelan como enlaces rígidos conectando vecinos hélices que se colocan inicialmente en un panal o un enrejado de cuadrados, proporcionando restricciones internas que deforman ADN desde su conformación recta, barra-como a formas complejas, como se muestra en detalle en [4, 7-8]. El modelo también explica el efecto de la columna vertebral los nicks en filamentos de la DNA, elasticidad entrópica de ADN utilizado para diseñar, por ejemplo, las estructuras de tensegridad [10] y cruces distantes para estructuras de alambre modelo [4]. Cada una de estas características del modelo es aproximada, sin embargo y por lo tanto sigue siendo un área activa de investigación. Predicción computacional de formas deformadas de ADN se realiza utilizando el método de elemento finito implementado en el programa de software comercial ADINA (ADINA R & D, Inc.), que es una técnica numérica bien establecida para el análisis de la compleja mecánica estructural y dinámica [11]. Las fluctuaciones inducida térmicamente de nanoestructuras de ADN se calculan usando el teorema de equipartición de la mecánica estadística y el análisis de modo normal, como se muestra para las proteínas en el [12-13]. Modelos atómicos de nanoestructuras de ADN se generan de las fluctuaciones térmicas y formas de solución 3D como se muestra en el diseño de recolección de luz nanodispositivos [14], molde de DNA para estructuras inorgánicas [15] y estructuras de ADN libre Enrejado [16].
Los resultante cadnano los archivos fueron subidos a CanDo para la simulación de cuerpo sólido con los siguientes parámetros.
CanDo volvió simulaciones de cuerpo sólido con un rango de desplazamiento final de entre 1,6 y 6,2 nanómetros. La simulación fue ilustrada por figuras y películas. En las primeras iteraciones de este diseño, la gama de dislocación excedió ~ 50 nanómetros (recordemos que hélices de ADN son alrededor de 2 nanómetros de diámetro, y el diseño entero aquí es alto 120nm y 60nm amplia en unirse a la A). Esto fue reducido grandemente por iterar la colocación de los crossovers, y nuestras simulaciones resultantes de CanDo demuestran que ninguna sola hélice o subunidad helicoidal pequeña muestra de grandes desplazamientos. Cualitativamente, un gran giro global se coloca en la estructura, pero resultados de Rothemund siguientes, se espera que los resultados experimentales siguen siendo planas en una superficie de mica en AFM.











